产品货号:F1501S
储存条件:-20℃
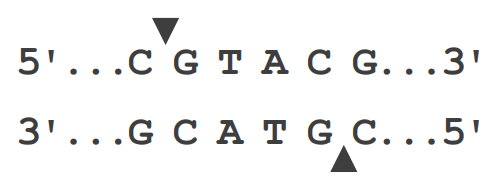
SgeI 可识别与切割含有 5-mC 位点的 DNA 靶标序列,一条链或双链甲基化均可被识别。
产品组成
组分 | 规格 |
LabFD Sgel (5 U/μl) | 50ul |
10×LabFD™ Buffer | 1ml |
产品简介
SgeI可切割在单链或双链DNA上含有5-甲基胞嘧啶的DNA靶标。SgeI限制性内切酶可识别m5CNNG(9/13)^位点,并于37℃下在其独特的缓冲液中的切割效果最佳。为确保一致的性能,该酶Storage Buffer中包含预混合BSA,其可增强酶的稳定性,并与DNA制剂中可能存在的污染物相结合。
酶活定义
在 1×LabFD™ Buffer 条件下,1μg pUC19-SgeI DNA (Dcm+) 在50μl 反应体系中37℃孵育1 h,不断增加酶量,直至酶切产物DNA带型不随着酶量的增加而发生变化,此时酶量定义为1 U。
质量控制
核酸内切酶残留检测
将 3U Sgel 与超螺旋质粒 DNA 在 37℃温育 4 h,通过DNA电泳检测质粒无变化。
核酸外切酶残留检测
将 5U Sgel 与双链 DNA 底物在 37℃温育 1 h,通过 DNA 电泳检测双链DNA 底物无变化。
注意事项
1. 底物至少需要 2 个 SgeI 识别序列才能有效酶切。
2. 甲基化 DNA 完全酶切取决于 SgeI 的识别位点的数量,另外由于识别位点酶切产生的DNA产物会促进SgeI 的非特异性酶切,因此建议酶切时优化SgeI酶量用于酶切反应。
使用方法
1. DNA 酶切流程
①在冰上按如下建议的加样顺序配制反应体系:
DNA | |
ddH2O | to 20 μl |
10x LabFD™ Buffer | 2 μl |
底物 DNA | 2 μl (0.5~2 μg) |
Sgel | 0.2~1 μl |
Total | 20 μl |
注:反应体系可以按比例放大或缩小。反应时间不建议超过 1 h。
② 轻柔吸打或轻弹管壁以混匀(切勿涡旋),然后瞬时离心以收集挂壁液滴;
③ 37℃温育1 h;
④ 80℃温育 20 min即可使酶失活,停止反应(可选)。
甲基化修饰影响
Dam | Dcm | CpG | EcoKI | EcoBI
|
无影响 | 总是切割被 Dcm甲基转移酶甲基化的DNA | 切割与 CpG 甲基化序列重叠的靶点 | 无影响 | 无影响 |





